[배경]
유전체 염기서열에서 획득한 유전체 변이 정보인 Single Nucleotide Polymorphism 정보는 특정 개체 및 특정 품종에 따라 다른 변이 양상을 나타낼 수 있기 때문에 동일개체를 확인하거나,
동일 품종을 구분하는데 활용이 가능합니다. 따라서 이번 경진대회에서는 개체 정보와 SNP 정보를 이용하여 A, B, C 품종을 분류하는 최고의 품종구분 정확도를 획득하는 것이 목표입니다.
농축수산 현장에서는 유전체 변이정보를 이용해서 품종을 구분하는 연구를 통해 품종의 다양성 혹은 품종 부정유통을 방지하기 위해 많이 활용하게 됩니다.
[주제]
개체와 SNP 정보를 이용하여 품종 분류 AI 모델 개발
[설명]
시장에서 세 품종이 동시에 유통될 때, 각 품종의 고유한 생산품목(우유 및 식육)의 가치 및 가격 산정에 부정유통이 차단되기 위해 현장에서 사용 할 수 있는 AI 모델이 필요합니다.
즉, 많은 SNP 정보를 통해 분류하는 것보다, 보다 더 적은 SNP 정보로 높은 분류 성능을 내는 것이 중요합니다.
따라서 이번 경진대회에서는 개체 정보와 사전에 구성된 15개의 SNP 정보를 바탕으로 품종 분류 모델을 개발해야 합니다.
[주최 / 주관]
- 주최: 충남대학교 바이오AI융합연구센터, 티엔티리써치, AI Frenz
- 주관: 데이콘
[결과]

[코드]
Import
import os
import time
import glob
import random
import numpy as np
import pandas as pd
import warnings
warnings.filterwarnings('ignore')
import lightgbm as lgb
from lightgbm import LGBMClassifier
# sklearn
from sklearn.metrics import log_loss, accuracy_score, classification_report
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import StratifiedKFold,StratifiedShuffleSplit, train_test_split, cross_val_score
from sklearn.preprocessing import OneHotEncoder, LabelEncoder
# optuna
import optuna
from optuna.samplers import TPESampler
from optuna.pruners import SuccessiveHalvingPruner
# tensorflow
import tensorflow as tf
from tensorflow import keras
import tensorflow_addons as tfa
from keras.utils import to_categorical
from keras.models import Sequential
from keras.layers import Dense, Dropout
from keras.callbacks import ReduceLROnPlateau, ModelCheckpointData Load / Preprocessing
train = pd.read_csv('./data/train.csv')
test = pd.read_csv('./data/test.csv')
train = train.drop(columns=['id', 'father', 'mother', 'gender'])
test = test.drop(columns=['id', 'father', 'mother', 'gender'])
TARLE = LabelEncoder()
SNPLE = LabelEncoder()
SNPCOL = [f'SNP_{str(x).zfill(2)}' for x in range(1,16)]
snp_data = []
for col in SNPCOL:
snp_data += list(train[col].values)
train.iloc[:,-1] = TARLE.fit_transform(train.iloc[:,-1])
SNPLE.fit(snp_data)
for col in train.columns:
if col in SNPCOL:
train[col] = SNPLE.transform(train[col])
test[col] = SNPLE.transform(test[col])EDA
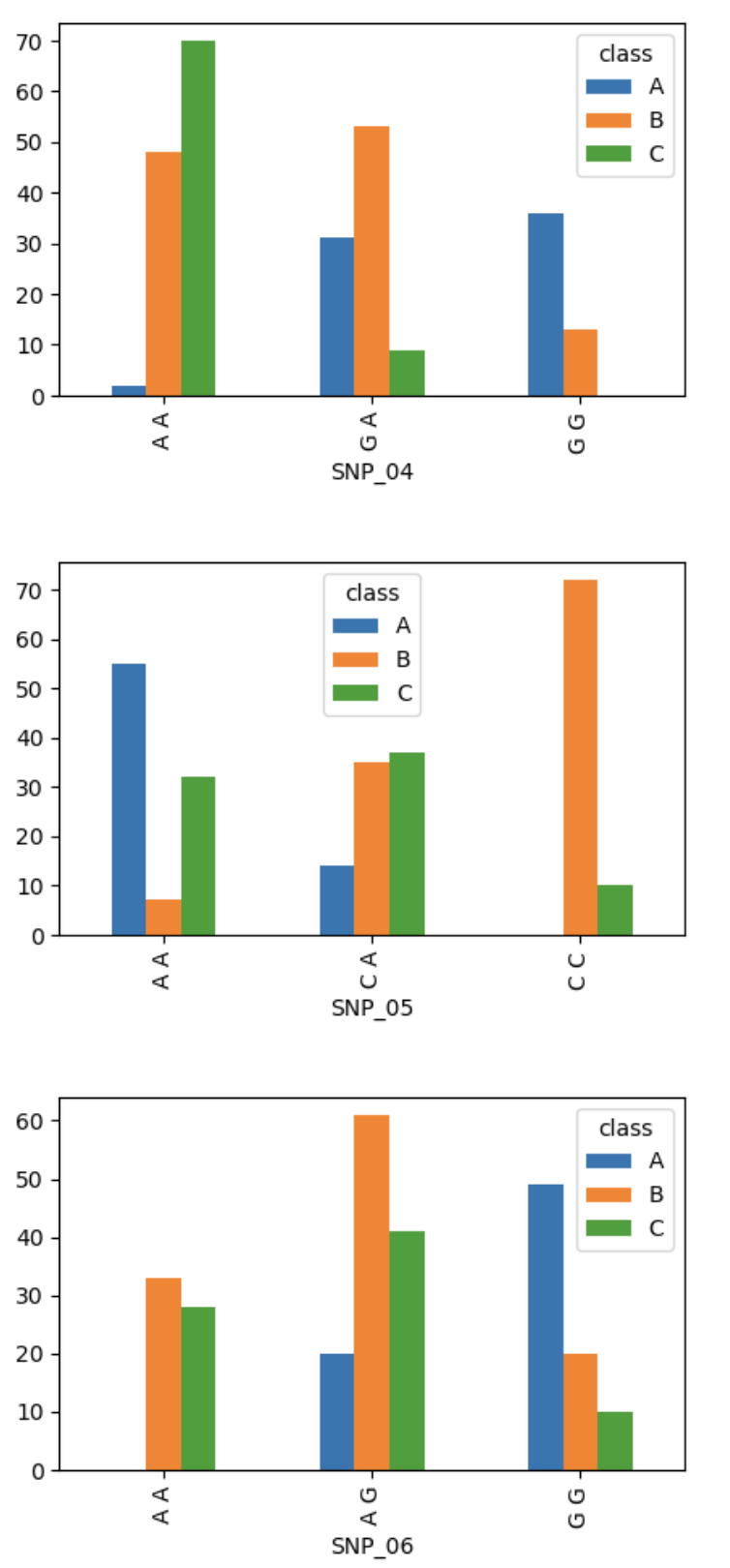
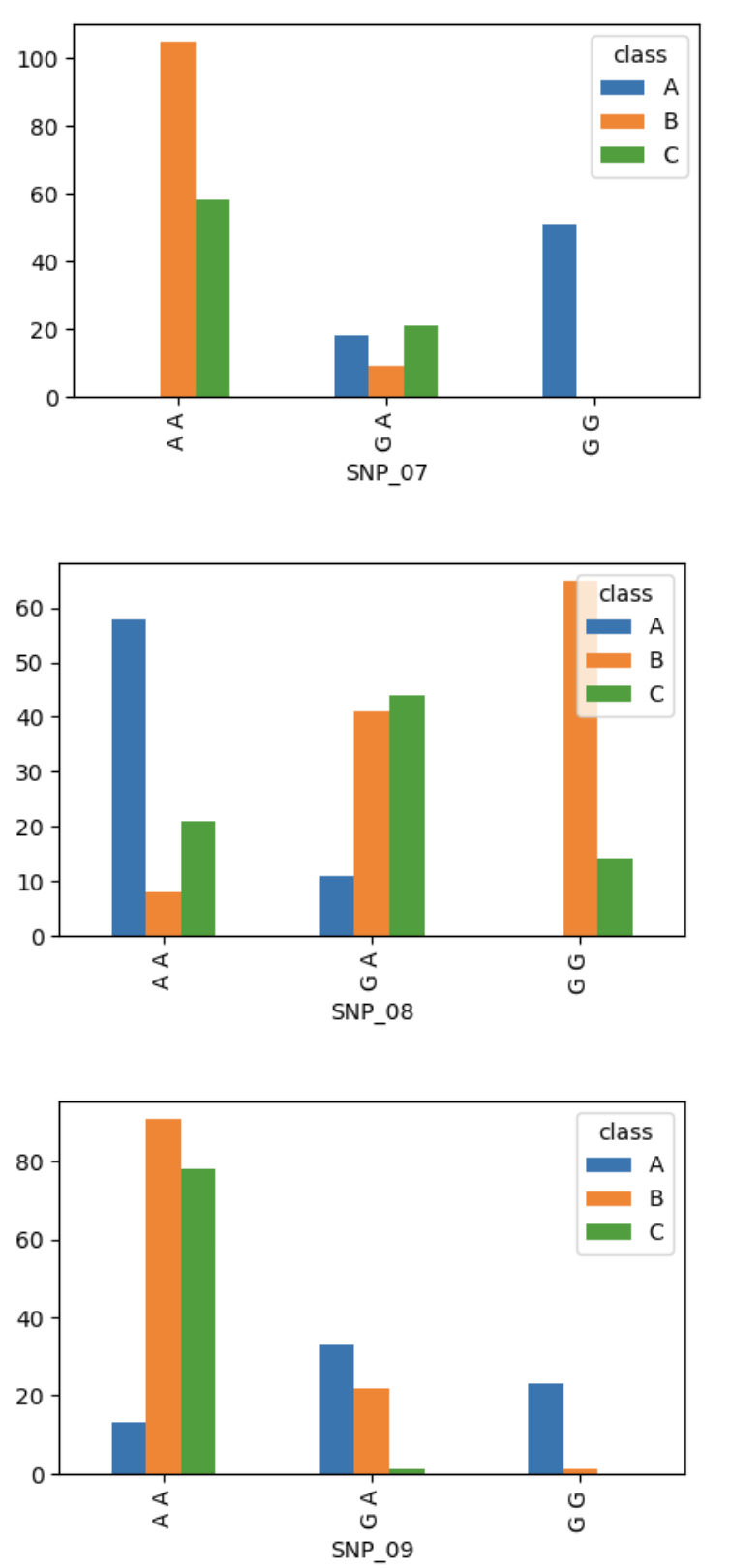

StratifiedShuffleSplit
- 데이터 불균형을 해소하기 위해 사용
# provides train/test indices to split data in train/test sets.
split = StratifiedShuffleSplit(n_splits=1, test_size=0.2, random_state=42)
for train_idx, test_idx in split.split(train, train['trait']):
x_train = train.iloc[:,:-1].loc[train_idx]
x_test = train.iloc[:,:-1].loc[test_idx]
y_train = train.iloc[:,-1].loc[train_idx]
y_test = train.iloc[:,-1].loc[test_idx]
# y_train = to_categorical(y_train)
# y_test = to_categorical(y_test)Optuna
def objective(trial):
params = {
"objective": "multiclass",
"metric": "multi_logloss",
"verbosity": -1,
"boosting_type": "gbdt",
"num_class": 3,
"lambda_l1": trial.suggest_float("lambda_l1", 1e-8, 10.0, log=True),
"lambda_l2": trial.suggest_float("lambda_l2", 1e-8, 10.0, log=True),
"num_leaves": trial.suggest_int("num_leaves", 2, 256),
"feature_fraction": trial.suggest_float("feature_fraction", 0.4, 1.0),
"bagging_fraction": trial.suggest_float("bagging_fraction", 0.4, 1.0),
"bagging_freq": trial.suggest_int("bagging_freq", 1, 7),
"min_child_samples": trial.suggest_int("min_child_samples", 5, 100),
}
score = []
clf = LGBMClassifier(**params)
clf.fit(x_train, y_train)
pred = clf.predict(x_test)
score = accuracy_score(y_test, pred)
return score
# Hyperparameter Tuning
study = optuna.create_study(direction='maximize', sampler=TPESampler(seed=0), pruner=SuccessiveHalvingPruner())
study.optimize(objective, n_trials=2000)Train
clfs = []
clf = LGBMClassifier(**study.best_params)
clf.fit(x_train, y_train)
clfs.append(clf)Soft Voting
submit = pd.read_csv('./data/sample_submission.csv')
pred = [clf.predict_proba(test) for clf in clfs]
pred = np.mean(pred, axis=0)
submit['class'] = TARLE.inverse_transform(np.argmax(pred, axis=1))Submission
submit.to_csv('./zzgr_submission.csv', index=False)[후기]
이번 대회를 하면서 sklearn에 내장된 모든 Classifier 및 Boosting계열 모델, DNN 등을 사용해봤다. 열댓개의 모델을 사용해보고 괜찮은 성능을 낸 모델들에 한해서 GridSearchCV와 Optuna를 적용해봤다. 그런데 성능이 전혀 개선되지 않았고, 모델링의 문제가 아니라 데이터 셋이 너무 작아서 생긴 문제라고 생각이 들었다. 더불어 EDA결과 클래스 불균형이 있는걸로 판단하여 StratifiedShuffleSplit를 이용하여 데이터 불균형을 해소하려고 했다. 그러나 대회가 끝나고 다른 참가자 코드를 보면서 불균형을 해소한 것이 아니라 불균형한 비율을 맞춰준 꼴이었다. 개멍청... Public 10등 정도를 유지해서 입상이라도 할 줄 알았는데, 웬걸 Private 36등으로 끝났다.
1등 팀의 경우에는 SNP_info에 있는 chrom, cm 등등을 Clustering해서 특징 별로 SNP 결합도 해보았고 여러 파생변수를 만들어서 최종 80여개 정도 만들었고 Feature Selection을 진행하여 최종 피처셋을 결정했다. 데이터 불균형에 대한 해결 방법은OverSampling, UnderSampling 및 모델에서 class_weight, sample_weight를 조절하는 등 여러 실험 결과 BorderlineSMOTE를 사용했다.
보고 많이 배웠다... 다음엔 더 잘해야지!
'📂 프로젝트 > ◾ DACON' 카테고리의 다른 글
| [데이콘/DACON] 상추의 생육 환경 생성 AI 경진대회 (낯선 데이터 다루기, 데이터 전처리, 시계열 데이터 처리, EDA, GAN) (0) | 2023.02.08 |
|---|

댓글